L’ ATP fornisce energia al lavoro cellulare accoppiando reazioni esoergoniche a reazioni endoergoniche
Definizione
Tutte le cellule modulano l’attività dei propri geni per adattarsi ai mutamenti dell’ambiente; nelle specie pluricellulari, tuttavia, questa capacità è stata raffinata fino a sostenere un’elevata specializzazione, con la generazione ordinata di molteplici tipi cellulari differenziati. Una caratteristica cruciale di tale processo è la stabilità delle decisioni di destino: quando una cellula intraprende un percorso di differenziamento, la configurazione di espressione genica che ne deriva tende a persistere lungo le divisioni successive. Ne consegue che segnali anche transitori devono imprimere una modifica duratura, un “ricordo” molecolare, affinché l’identità cellulare rimanga coerente nel tempo e i tessuti mantengano la loro organizzazione e funzione.
Per contro, variazioni di espressione più semplici, sia nei procarioti sia negli eucarioti unicellulari, sono spesso effimere. Un esempio classico è il repressore del triptofano nei batteri: la sua azione inibitoria sull’operone del triptofano si manifesta solo in presenza dell’amminoacido; quando il triptofano viene rimosso dal terreno, i geni si riattivano e le cellule figlie non conservano traccia dell’esposizione precedente. Negli organismi pluricellulari, invece, la memoria cellulare emerge da meccanismi che includono feedback trascrizionali, modificazioni epigenetiche della cromatina e, in certi casi, l’ereditarietà mitotica di stati strutturali del cromosoma. Queste strategie, congiuntamente, permettono di generare, consolidare e trasmettere nel tempo identità cellulari distinte in organismi complessi come nematodi, insetti e mammiferi.
Tra i processi che favoriscono tale memoria si annoverano:
- circuiti di autoregolazione o di feedback positivo dei regolatori della trascrizione, che stabilizzano lo stato di espressione prescelto;
- modificazioni covalenti degli istoni e rimodellamento della cromatina, che modulano l’accessibilità del DNA in modo propagabile;
- metilazione del DNA in specifici contesti genomici, associata a silenziamento stabile;
- associazioni nucleari di lunga durata, come domini topologici e interazioni enhancer-promotore mantenute attraverso le divisioni.
I sistemi finora considerati in ambito batterico sono spesso governati da pochi regolatori della trascrizione che esercitano effetti marcati su singoli operoni. Negli eucarioti, al contrario, la maggior parte dei geni è regolata da insiemi numerosi di proteine che agiscono in concerto, integrando molteplici segnali per ottenere un’espressione spazialmente e temporalmente appropriata. Con il termine controllo combinatorio si indica proprio la logica con cui più regolatori cooperano per definire il profilo di espressione di un gene.
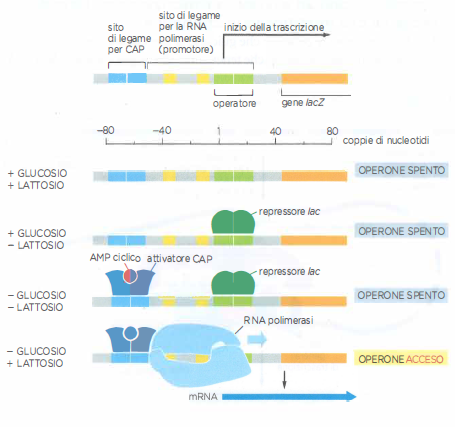
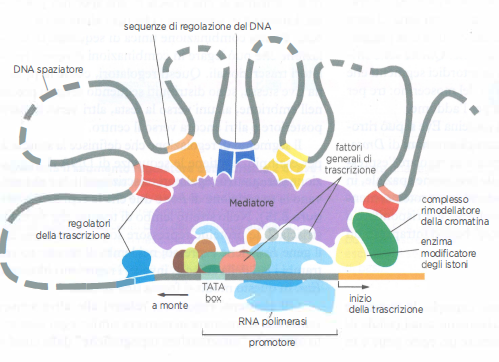
Un riferimento didattico per comprendere l’integrazione di segnali è l’operone lac batterico (Figura 03.11-12), che risponde a due input distinti. Nei genomi eucariotici, tuttavia, la complessità cresce: i geni sono modulati da dozzine di fattori che riconoscono sequenze regolatrici distribuite su intervalli che vanno da poche decine a molte migliaia di coppie di basi, includendo promotori, enhancer distali e, talvolta, elementi intronici. Queste proteine, una volta reclutate, cooperano nel promuovere l’assemblaggio del complesso del Mediatore, il quale funge da piattaforma integrativa capace di coordinare rimodellatori della cromatina, enzimi che modificano gli istoni, l’RNA polimerasi II e i fattori generali della trascrizione (Figura 03.11-01).
Il risultato nasce dall’equilibrio dinamico tra attivatori e repressori, la cui combinazione determina la probabilità di innesco della trascrizione, la sua intensità e la precisione temporale. Interazioni cooperative fra regolatori che occupano siti adiacenti possono conferire comportamenti simili a porte logiche (per esempio funzioni assimilabili a AND/OR), mentre la presenza di cofattori e la struttura tridimensionale della cromatina, con loop mediati da proteine architetturali, consentono a elementi distali di comunicare efficacemente con i promotori bersaglio. In tal modo, la cellula integra segnali estrinseci (ormoni, fattori di crescita, stress) e intrinseci (stato del ciclo, metabolismo) per generare risposte proporzionate e specifiche al tipo cellulare.
La traduzione operativa di questo schema comprende:
- selezione di combinazioni di siti di legame nel DNA, con affinità e specificità variabili;
- reclutamento sequenziale o sinergico di coattivatori e corepressori, inclusi acetiltransferasi, deacetilasi, metiltransferasi e complessi di rimodellamento della cromatina;
- formazione di domini cromatinici permissivi o repressi, la cui persistenza contribuisce alla memoria di espressione;
- integrazione con segnali nucleari di organizzazione spaziale, che favoriscono il contatto enhancer–promotore.
Oltre a modulare singoli geni, le cellule devono coordinare rapidamente gruppi di geni funzionalmente correlati. Nei batteri tale coordinazione è spesso ottenuta raggruppando geni in un unico operone, sotto il controllo di un singolo promotore (Figura 03.11-13). Nelle cellule eucariotiche, dove ciascun gene possiede il proprio promotore e set di elementi regolatori, la sincronizzazione avviene sfruttando elementi di risposta condivisi e il controllo combinatorio: l’aggiunta o la rimozione di un particolare regolatore può “completare” l’insieme necessario per l’attivazione o la repressione di più geni contemporaneamente, analogamente a un codice che, inserito nella combinazione corretta, rende operativo l’intero meccanismo.
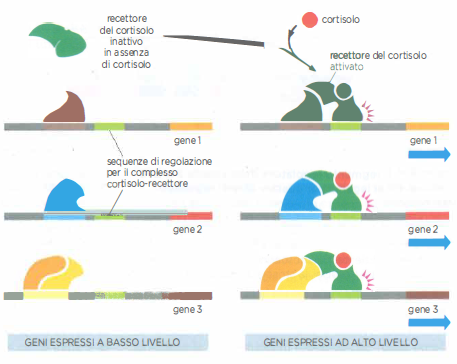
Quando differenti geni contengono siti di legame per il medesimo regolatore, la presenza di tale proteina consente di orchestrare la loro espressione come un’unità coordinata, pur mantenendo per ciascun gene specificità addizionali fornite da cofattori e contesto cromatinico. Nell’essere umano, un caso esemplare è la risposta al cortisolo: il recettore del cortisolo, una volta complessato all’ormone, migra nel nucleo e si lega a sequenze regolatrici presenti in molteplici geni bersaglio, in particolare nelle cellule epatiche, favorendo programmi che sostengono la gluconeogenesi e l’adattamento a digiuno prolungato o stress (Figura 03.11-02). Al diminuire della concentrazione dell’ormone, il complesso si dissocia e l’espressione rientra ai livelli basali.
Questo principio è ampiamente generalizzabile. Un ulteriore esempio è fornito dalla risposta all’ipossia: il fattore HIF-1, stabilizzato quando l’ossigeno è scarso, si lega a elementi di risposta all’ipossia distribuiti nel genoma e attiva coortamente geni coinvolti in glicolisi, angiogenesi e trasporto di ossigeno. Sebbene i dettagli dei cofattori differiscano a seconda del tessuto e del contesto, il meccanismo rimane lo stesso: un singolo regolatore, agendo su elementi di risposta condivisi, sincronizza l’attività di molteplici geni producendo un effetto fisiologico integrato.
Dal punto di vista molecolare, la coordinazione efficiente richiede:
- motivi di legame ripetuti o conservati in insiemi di geni funzionalmente correlati;
- affinità di legame modulata dalla cromatina e dall’occupazione di cofattori, così da conferire gradazioni di risposta;
- integrazione con segnali post-traduzionali sul regolatore stesso, come fosforilazione o legame a ligandi, che ne controllano localizzazione e attività;
- decadimento regolato del segnale (per esempio rimozione dell’ormone o degradazione del fattore), per garantire reversibilità e prevenire risposte inappropriate.
In sintesi, anche in un quadro di controllo combinatorio complesso, un singolo regolatore può agire come nodo decisionale capace di accendere o spegnere interi programmi trascrizionali, assicurando al contempo specificità tissutale e flessibilità temporale.
Negli eucarioti, un numero limitato di regolatori della trascrizione, agendo in combinazione, è in grado di orchestrare programmi di espressione che coinvolgono migliaia di geni. Questo principio, fondamentale per l’omeostasi tissutale, è ancora più rilevante durante l’embrionogenesi, quando cellule inizialmente simili intraprendono percorsi di differenziamento divergenti. Un caso emblematico è la miogenesi scheletrica nei mammiferi, in cui la specificità cellulare emerge dalla co‑attivazione di ampi repertori di geni che codificano componenti dell’apparato contrattilo e dell’eccitabilità di membrana. Tra le caratteristiche che distinguono la fibra muscolare scheletrica si annoverano:
- isoforme muscolo-specifiche di actina e miosina che costituiscono i sarcomeri;
- canali ionici voltaggio-dipendenti e recettori di membrana che conferiscono la risposta allo stimolo neuronale;
- proteine strutturali e regolative del citoscheletro e della giunzione neuromuscolare;
- enzimi e trasportatori dedicati al metabolismo energetico ad alta richiesta.
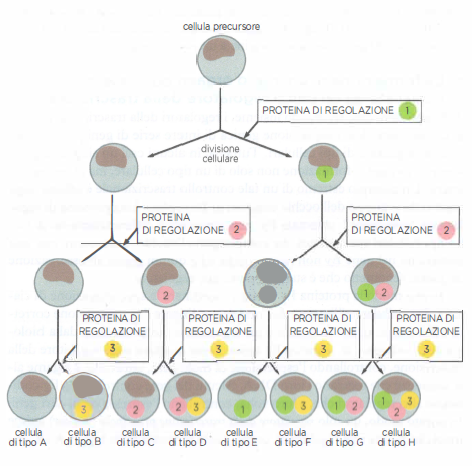
La comparsa coordinata di questi prodotti genici coincide con l’attivazione, nelle cellule progenitrici, di specifici regolatori della trascrizione che riconoscono sequenze cis-regolatrici (promotori ed enhancer) dei geni muscolo-specifici. Nei vertebrati, fattori quali i membri della famiglia MRF (ad esempio MyoD, Myf5, Myogenin) e complessi cooperanti come MEF2 costituiscono nodi chiave del programma miogenico, benché il principio sia generale e valga per molti altri destini cellulari. Analogamente, insiemi distinti di regolatori definiscono le identità neuronale, epiteliale, ematopoietica, e così via. Uno schema del modo in cui combinazioni diverse di regolatori indirizzano lo sviluppo verso tipi cellulari differenti è riportato nella (Figura 03.11-03). Alcuni regolatori, al contrario, sostengono uno stato progenitoriale indifferenziato, come illustrato per i precursori nella (Figura 03.11-03).
Alcune cellule indifferenziate possiedono una versatilità straordinaria: le cellule staminali embrionali (ES) sono pluripotenti, ossia capaci di generare tutti i tipi cellulari somatici. La transizione verso uno specifico destino comporta un riposizionamento su larga scala del profilo trascrizionale: i geni funzionali per il nuovo tipo cellulare vengono potenziati, mentre interi moduli genici alternativi sono attenuati o silenziati. In genere, un singolo regolatore della trascrizione può modulare l’attività di centinaia, talora migliaia, di loci (Figura 03.11-04). Poiché ciascun gene è controllato da molteplici fattori e moduli regolativi, un repertorio relativamente contenuto di regolatori, combinato in schemi diversi, è sufficiente a costruire reti di regolazione di enorme complessità in grado di generare l’intero spettro delle identità cellulari. Stime correnti suggeriscono che circa 1000 regolatori della trascrizione possano coordinare l’espressione dei ~24 000 geni che concorrono alla formazione di un organismo umano.
La dimensione combinatoria non riguarda solo la presenza dei fattori, ma anche l’accessibilità del cromatina e la logica dei moduli enhancer: fattori “pioneer” possono aprire regioni compatte, consentendo ad altri regolatori di legarsi; ulteriori interazioni cooperative o antagonistiche definiscono soglie e temporalità dell’attivazione. Questi principi, agendo in sinergia, rendono conto della precisione e della robustezza dei programmi di differenziamento.
Sebbene nella maggior parte dei casi la specificazione cellulare emerga da combinazioni di regolatori, in alcune circostanze un singolo “regolatore principale” è sufficiente ad avviare lo sviluppo coordinato di un intero organo. Un caso paradigmatico è lo sviluppo dell’occhio in Drosophila: la proteina Ey (Eyeless) può scatenare il differenziamento concertato dei molteplici tipi cellulari che compongono il tessuto oculare. Mutazioni inattivanti nel gene Ey producono moscerini privi di occhi, fenomeno che ha permesso di identificare il ruolo cardine di questo regolatore. Ey funziona in modo analogo ad altri fattori trascrizionali: si lega a sequenze regolatrici del DNA e attiva una batteria di geni, inclusi altri regolatori che propagano il segnale lungo una rete di controllo multilivello.
Questa architettura di rete, schematizzata nella (Figura 03.11-04), genera una cascata di eventi in cui nodi regolatori secondari coordinano moduli genici specifici, assicurando l’emergere, nel giusto ordine e nelle corrette proporzioni, di molteplici identità cellulari e della loro disposizione spaziale tridimensionale. La potenza di un regolatore principale è tale da attivare il programma oculare anche in sedi ectopiche: l’espressione sperimentale di Ey in regioni embrionali destinate a formare appendici ha prodotto occhi ectopici, per esempio nel segmento di una zampa (Figura 03.11-05). Questo risultato illustra come reti regolative opportunamente innescate possano ricapitolare interi programmi organogenetici al di fuori del loro contesto nativo.
Concetti analoghi valgono in altri sistemi: l’omologo vertebrato di Ey, PAX6, è essenziale per l’occhio dei mammiferi; in più, nei distretti ematopoietici fattori quali GATA1 o PU.1 possono fungere da interruttori di linea, stabilendo bias di destino attraverso reti con circuiti di attivazione e repressione incrociata.
L’osservazione che Ey possa indurre la formazione di un occhio in una sede anomala in un embrione riflette la cooperazione di molti regolatori in un ambiente embrionale ricco di segnali. Sorprendentemente, anche in coltura un set appropriato di regolatori della trascrizione può riprogrammare cellule già differenziate verso un’identità alternativa. L’introduzione artificiale del gene che codifica MyoD, per esempio, è in grado di convertire fibroblasti cutanei coltivati in cellule simil‑muscolari, evidenziando come questi fibroblasti, condividendo l’origine mesodermica, posseggano gran parte dei co-regolatori necessari al programma miogenico e come l’aggiunta di MyoD completi la combinazione richiesta.
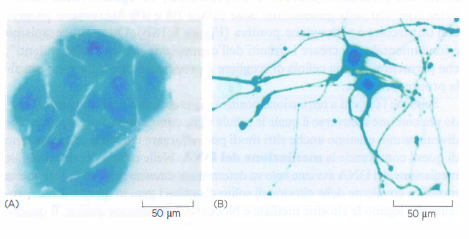
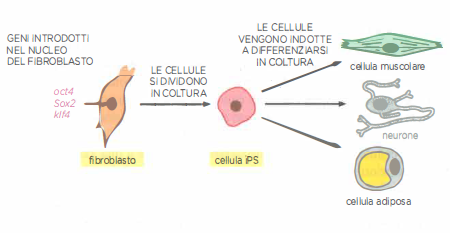
Trasformazioni ancora più marcate sono state ottenute attivando programmi neuronali: un insieme di regolatori specifici per i neuroni, espresso in cellule epatiche in coltura, ne promuove la conversione in neuroni funzionali (Figura 03.11-06). Un’ulteriore applicazione è la “de‑differenziazione” controllata: la combinazione di regolatori rappresentata nella (Figura 03.11-04) consente la generazione di cellule staminali pluripotenti indotte (iPS), che condividono con le cellule ES proprietà chiave quali pluripotenza e auto-rinnovamento e possono essere guidate verso molteplici identità specializzate (Figura 03.11-07). Questo approccio, inizialmente messo a punto in fibroblasti, è stato esteso a numerosi tipi cellulari umani.
Le cellule derivate da iPS sono oggi impiegate nello studio di patologie e in protocolli terapeutici sperimentali. Accanto a questi esempi, si sono ottenute conversioni dirette tra tipi cellulari senza passare per uno stato staminale (transdifferenziamento), come l’induzione di neuroni da fibroblasti mediante fattori quali Ascl1, Brn2 e Myt1l, o la conversione mirata di cellule esocrine pancreatiche in cellule beta con combinazioni di Pdx1, Ngn3 e Mafa. Questi risultati rafforzano l’idea che, identificando la corretta combinazione di regolatori, sia possibile produrre in laboratorio molti tipi cellulari di interesse.
Una volta stabilita, l’identità cellulare tende a mantenersi nel tempo e nelle generazioni cellulari. Alcune cellule altamente specializzate, come i neuroni e le fibre muscolari scheletriche, sono terminalmente differenziate e cessano di proliferare; molte altre, come fibroblasti, cellule muscolari lisce ed epatociti, conservano capacità replicative pur mantenendo stabile la propria identità. La continuità di identità, detta memoria cellulare, implica la trasmissione alle cellule figlie dei pattern di espressione genica che definiscono il tipo cellulare. Nel contesto illustrato nella (Figura 03.11-03), la produzione di regolatori chiave, una volta avviata, deve proseguire nelle progenie.
Un meccanismo semplice e potente è il circuito a retroazione positiva, in cui un regolatore cardinale attiva, oltre ai geni effettori, anche la trascrizione del proprio gene. A ogni divisione, la proteina regolatrice viene ripartita tra le figlie e continua a sostenere il circuito autostimolante, garantendo persistenza dell’espressione (Figura 03.11-08). La proteina Ey e i regolatori coinvolti nella generazione di ES e iPS si inseriscono in tale logica circuitale (Figura 03.11-04), creando “moduli indipendenti” di espressione genica che stabilizzano le decisioni di destino.
Accanto alla retroazione positiva, operano ulteriori meccanismi epigenetici che consolidano l’identità:
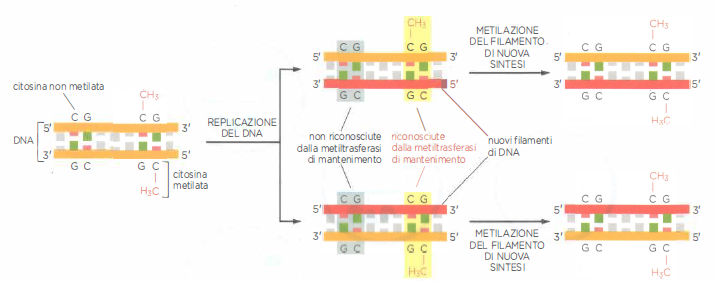
- Metilazione del DNA: nei vertebrati, la metilazione avviene prevalentemente su citosine in dinucleotidi CpG (Figura 03.11-09) e tende a reprimere la trascrizione reclutando proteine leganti il DNA metilato e complessi rimodellatori di cromatina; un enzima di mantenimento riconosce le emieliche metilate dopo la replicazione e copia il pattern sul filamento neosintetizzato, preservando la metilazione parentale (Figura 03.11-10);
- Modificazioni degli istoni: durante la duplicazione del DNA, ciascuna doppia elica figlia riceve circa metà degli istoni parentali, che portano segni covalenti (per esempio metilazioni o acetilazioni). Complessi “lettore‑scrittore” riconoscono tali segni sugli istoni ereditati e propagano le stesse modificazioni sui nuovi istoni vicini, ristabilendo lo stato della cromatina della cellula madre (Figura 03.11-11);
- Complessi regolatori della cromatina: sistemi come le famiglie Polycomb e Trithorax contribuiscono, rispettivamente, al mantenimento di stati repressi o attivi su loci chiave dello sviluppo, agendo in modo concorde con i segni istonici;
- Architettura nucleare: l’organizzazione tridimensionale del genoma e l’ancoraggio a domini nucleari specifici possono stabilizzare programmi trascrizionali, sostenendo contatti enhancer‑promotore favorevoli o sfavorevoli alla trascrizione.
Poiché tali processi trasmettono informazioni regolative senza modificare la sequenza nucleotidica, rientrano nell’eredità epigenetica. La loro azione congiunta consente di convertire segnali transitori in cambiamenti durevoli dell’espressione genica, con conseguenze cruciali per lo sviluppo, la rigenerazione tissutale e la patogenesi di numerose malattie.