In seguito all’ossidazione del piruvato il ciclo dell’acido citrico completa l’ossidazione delle molecole organiche con liberazione di energia
Definizione
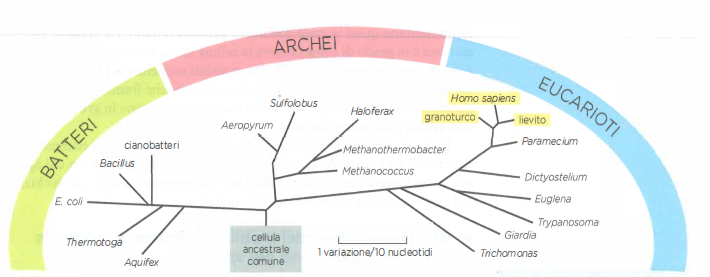
L’albero della vita raffigurato in (Figura 04.04-08) riunisce linee evolutive tra loro lontane, dai cianobatteri, che immettono ossigeno nell’atmosfera, agli animali, compreso l’uomo, che sfruttano l’ossigeno per la respirazione e per sostenere il metabolismo. In tale rappresentazione non compaiono, tuttavia, entità peculiari collocate ai margini della definizione convenzionale di “vivente”: elementi genetici parassitari e virus. Pur essendo costituiti dagli stessi acidi nucleici che caratterizzano tutte le forme di vita e pur disponendo di strategie per replicarsi e trasferirsi, questi sistemi non soddisfano pienamente i criteri biologici attribuiti agli organismi, come il metabolismo autonomo o la capacità di riproduzione indipendente.
La loro ubiquità e le loro modalità di azione li rendono, nondimeno, determinanti nell’evoluzione dei genomi e nella fisiopatologia umana. Gli elementi genetici mobili, detti colloquialmente geni saltatori, sono rilevabili in quasi tutte le cellule eucariotiche e procariotiche; nell’uomo, sequenze riconducibili a tali elementi occupano una frazione cospicua del genoma, pari a quasi la metà. La maggior parte di essi resta confinata nella cellula ospite, ma è capace di inserirsi in numerose posizioni del DNA nucleare o plasmidico. I virus, al contrario, sono pacchetti di materiale genetico racchiusi in un rivestimento proteico e talora lipidico, progettati per uscire da una cellula e colonizzarne altre, avviando cicli infettivi. Nel seguito vengono delineati la struttura e i meccanismi d’azione di questi sistemi, nonché le conseguenze sulla regolazione dell’espressione genica, sulla riorganizzazione ed espansione dei genomi e sulla dinamica della trasmissione delle malattie.
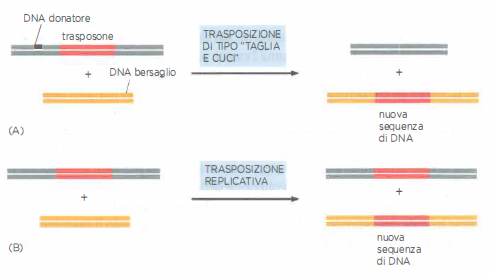
Gli elementi genetici mobili, o trasposoni, si classificano in primo luogo in base alla via con cui si spostano (trasposizione). Nei batteri prevalgono i trasposoni a solo DNA (DNA-only transposons), così chiamati perché la fase mobile è rappresentata dal DNA stesso, senza intermedio a RNA. All’interno di questa categoria si distinguono due modalità principali: il meccanismo “taglia e cuci” e la trasposizione replicativa. Nel primo caso, l’elemento viene escisso dal sito di origine e reinserito in un nuovo locus; nel secondo, l’elemento si duplica e una copia si integra altrove, lasciando intatta la sequenza originale nel punto di partenza (Figura 04.04-01).
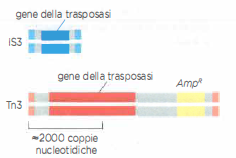
Ciascun elemento mobile codifica tipicamente un enzima chiave, la trasposasi, che orchestri le fasi di riconoscimento, taglio e integrazione. La trasposasi identifica sequenze cis-regolative specifiche presenti all’estremità del trasposone, spesso ripetizioni invertite terminali, e genera brevi duplicazioni del sito bersaglio al momento dell’inserzione, una firma molecolare frequente di questi eventi. In molti casi, oltre al gene per la trasposasi, i trasposoni trasportano geni accessori che conferiscono vantaggi selettivi in particolari contesti. Di rilievo clinico sono i trasposoni che veicolano geni di resistenza agli antibiotici: la loro mobilità ha facilitato la diffusione di resistenze in popolazioni batteriche diverse e anche il passaggio tra specie mediante elementi coniugativi o plasmidi, come illustrato in (Figura 04.04-02).
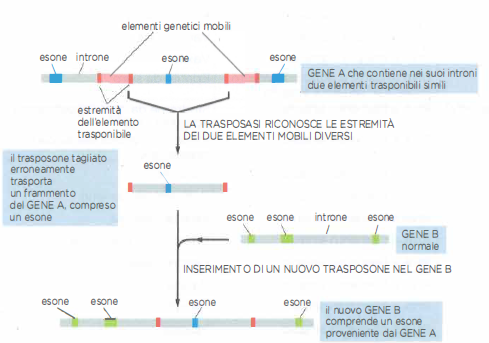
Oltre a spostarsi, i trasposoni possono rimodellare il genoma ospite. Se due elementi riconosciuti dalla stessa trasposasi si dispongono in prossimità sul medesimo cromosoma, la ricombinazione a carico delle loro sequenze terminali può portare alla rimozione dell’intervallo compreso e alla sua successiva integrazione in un’altra sede cromosomica o in un gene diverso (Figura 04.04-03). Tali riarrangiamenti, pur essendo talora deleteri, costituiscono anche un potente motore di innovazione genetica negli eucarioti, in quanto:
- possono interrompere o modificare geni esistenti, generando nuovi alleli e talora nuove funzioni proteiche mediante riassetti esonici;
- possono trasferire sequenze regolative, come promotori o enhancer, accanto a geni preesistenti, alterandone tempistica e livello di espressione;
- possono indurre duplicazioni segmentali, offrendo materia prima per la divergenza funzionale di copie geniche;
- possono favorire ricombinazioni ectopiche tra copie omologhe disperse, con conseguenti inversioni, delezioni o traslocazioni cromosomiche.
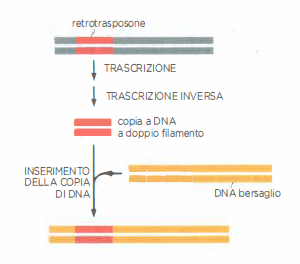
Accanto ai trasposoni a solo DNA, nei genomi eucariotici è ampiamente rappresentata una seconda grande classe, gli elementi a RNA (retrotrasposoni), che si spostano tramite copia a RNA seguita da retrotrascrizione in DNA; benché non dettagliati in (Figura 04.04-01), questi contribuiscono in misura sostanziale alla massa genomica umana e comprendono, tra gli altri, LINE e SINE. La persistenza e l’amplificazione di tali elementi spiegano perché, nell’uomo, le sequenze derivate da elementi mobili costituiscano una porzione considerevole del genoma.
L’impatto biologico degli elementi mobili va inquadrato in una prospettiva ecologica del genoma. Da un lato, la loro attività rappresenta una fonte di mutazione inserzionale e di instabilità, con possibili effetti patogeni se l’integrazione avviene in geni essenziali o in regioni regolative critiche; dall’altro, nel lungo periodo essi fungono da serbatoio di innovazione: promotori e siti di legame per fattori di trascrizione derivati da elementi mobili sono stati cooptati in reti regolative, e intere proteine hanno incorporato moduli esonici mobilitati in passato. Nei batteri, inoltre, la cooperazione tra trasposoni, plasmidi e isole genomiche accelera il trasferimento orizzontale di geni, inclusi quelli per la virulenza e il metabolismo di composti xenobiotici.
In sintesi, la capacità degli elementi genetici mobili di codificare gli enzimi necessari al proprio movimento, di replicarsi o di ricombinarsi con sequenze affini e di trasportare geni accessori, li rende attori centrali della plasticità genomica. Gli effetti che ne derivano spaziano dall’alterazione immediata dell’espressione genica alla ristrutturazione profonda dei cromosomi, con ripercussioni che vanno dall’adattamento evolutivo alla comparsa di resistenze antimicrobiche e a specifiche patologie.
Il confronto sistematico di numerosi genomi umani ha rivelato un risultato inatteso: una porzione considerevole del nostro DNA deriva da elementi genetici capaci di spostarsi. Questi “ospiti” costituiscono quasi metà del genoma e sono presenti in milioni di copie distribuite in tutto il DNA nucleare. Due modalità di mobilità sono rilevanti: da un lato i trasposoni a DNA, che si muovono con un meccanismo di “taglia e incolla” simile a quello schematizzato in (Figura 04.04-01); dall’altro i retrotrasposoni, che si propagano attraverso un intermedio a RNA, caratteristica tipica, per diffusione ed impatto, dei genomi eucariotici.
Tra i retrotrasposoni non-LTR, l’elemento L1 (o LINE-1, Long Interspersed Nuclear Element) rappresenta una famiglia abbondante. L1 viene trascritto dalle RNA polimerasi dell’ospite e codifica due proteine, note come ORF1p e ORF2p; quest’ultima possiede attività endonucleasica e di trascrittasi inversa. Dopo la traduzione, il complesso ribonucleoproteico L1 utilizza un processo detto “target-primed reverse transcription” per generare in situ una copia a DNA a doppio filamento dell’RNA trascritto, che può integrarsi in un nuovo sito genomico (Figura 04.04-04). Gli elementi L1 contribuiscono per circa il 15% al contenuto del genoma umano. Mentre poche copie rimangono potenzialmente attive, la grande maggioranza è ormai inattiva per accumulo di mutazioni deleterie.
Lo spostamento di retrotrasposoni può avere esiti patogeni nel singolo individuo, quando l’inserzione interrompe geni o regioni regolative. Sono stati documentati, ad esempio, eventi di inserzione L1 o ricombinazioni mediate da Alu che hanno innescato mutazioni in geni quali DMD, NF1 o APC, con conseguenze cliniche rilevanti. Un’altra famiglia estremamente proliferata è costituita dagli elementi Alu, retrotrasposoni non autonomi appartenenti alle SINE (Short Interspersed Nuclear Elements), derivati evolutivamente dall’RNA 7SL. Il genoma umano contiene circa un milione di copie Alu, per un totale vicino al 10% del DNA. Non codificando una propria trascrittasi inversa, Alu sfrutta il macchinario proteico di LINE-1 per retrotrascriversi e integrarsi.
Il confronto della distribuzione e delle subfamiglie di L1 e Alu in diversi mammiferi suggerisce una marcata espansione in tempi evolutivi recenti lungo la linea dei primati (Figura 04.03-03). L’accumulo di migliaia di siti di inserzione ha plasmato la regolazione genica tramite diversi meccanismi: fornitura di promotori e enhancers alternativi, creazione di nuovi siti di splicing, contributo a RNA non codificanti e riorganizzazioni cromosomiche. A livello di controllo dell’ospite, l’attività di questi elementi è in gran parte repressa da metilazione del DNA, modificazioni istoniche e piccole vie a RNA (ad esempio il sistema piRNA nelle cellule germinali), che limitano gli eventi di mobilità potenzialmente dannosi:
- Trasposoni a DNA: mobilità per “taglia e incolla”, mediata da trasposasi;
- Retrotrasposoni non-LTR: LINE (autonomi, es. L1) e SINE (non autonomi, es. Alu), con copia a RNA e reintegrazione via trascrittasi inversa;
- Retrotrasposoni LTR: strutture con ripetizioni terminali lunghe e geni omologhi a quelli retrovirali, oggi in gran parte inattivi nell’uomo.
La presenza massiccia e la dispersione di L1 e Alu suggeriscono che una quota non trascurabile dell’innovazione regolativa ed evolutiva nei primati sia stata influenzata da questi “parassiti molecolari”, i cui esiti, variando da effetti neutri a potenzialmente benefici o patogeni, hanno lasciato tracce profonde nei nostri cromosomi.
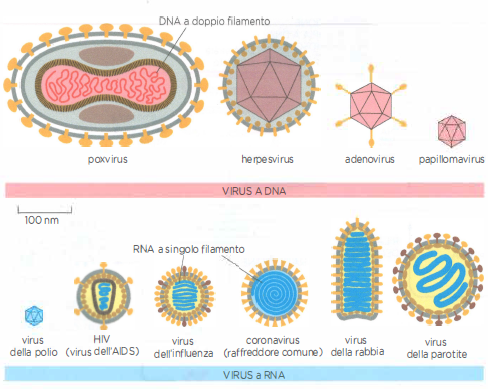
A differenza dei trasposoni, i virus formano particelle infettive capaci di uscire da una cellula, diffondersi e invadere nuove cellule o organismi. In termini strutturali, un virione è un genoma racchiuso in un rivestimento proteico (capside), talvolta avvolto da una membrana lipidica con glicoproteine virali. Per replicarsi, i virus devono penetrare in una cellula e sfruttarne i meccanismi per la sintesi di RNA, proteine e nuovi genomi. La maggior parte dei virus patogeni umani possiede genomi a DNA a doppio filamento o a RNA a singolo filamento (Tabella 04.04-01), sebbene esistano anche genomi virali a DNA a singolo filamento e a RNA a doppio filamento.
La dimensione genomica virale è estremamente variabile. I virus più piccoli codificano un numero ridotto di geni, anche solo tre, e sono racchiusi in capsidi formati dalla ripetizione di poche subunità proteiche identiche; quelli più complessi possono possedere centinaia di geni e un involucro articolato composto da molte proteine differenti (Figura 04.04-05). La limitata capacità di impacchettamento del capside impone una forte dipendenza dall’ospite: nessun virus codifica l’insieme completo di enzimi e fattori necessari alla propria moltiplicazione, e quindi il ciclo replicativo prevede l’appropriazione dei circuiti biosintetici cellulari (Figura 04.04-06). Tipicamente, il genoma virale codifica le proteine strutturali dell’involucro e fattori che modulano o dirottano le funzioni cellulari per ottimizzare replicazione e assemblaggio.
L’infezione può essere litica o persistente. Nelle infezioni litiche, l’accumulo della progenie virale porta spesso alla lisi della cellula infetta, con rilascio di nuove particelle che propagano l’infezione nelle cellule adiacenti; molti segni clinici sono riconducibili a questa citopatia, come nel caso di vescicole cutanee causate da virus a DNA che colpiscono l’epitelio. In altri contesti, i virus instaurano rapporti più duraturi, con latenza o cronica persistenza, in cui l’espressione genica virale è modulata per eludere le difese immunitarie e preservare la cellula ospite come serbatoio:
- Tipi di genoma virale: DNA o RNA, lineare o circolare, a singolo o doppio filamento, talvolta segmentato;
- Strategie di espressione: sintesi diretta di mRNA (+RNA), trascrizione da DNA virale, o impiego di polimerasi virali dipendenti da RNA;
- Esiti cellulari: lisi, latenza, persistenza, trasformazione, a seconda del virus e del contesto tissutale.
| Virus | Genoma | Patologia associata |
|---|---|---|
| Virus della varicella-zoster | DNA a doppio filamento | Varicella ed herpes zoster |
| Virus del morbillo | RNA a singolo filamento | Morbillo |
| Virus dell’epatite B | DNA parzialmente a doppio filamento e RNA intermedio | Epatite cronica e acuta da siero |
| Virus della rabbia | RNA a filamento singolo | Rabbia |
| Virus di Epstein-Barr (EBV) | DNA a doppio filamento | Mononucleosi infettiva |
| Virus della parotite | RNA a singolo filamento | Parotite |
| Virus della poliomielite | RNA a filamento singolo | Poliomielite |
| Virus influenzale di tipo A | RNA a filamento singolo | Influenza stagionale e complicanze respiratorie |
| Virus dell’herpes simplex | DNA a doppio filamento | Infezioni erpetiche ricorrenti |
| Virus dell’epatite C | RNA a singolo filamento | Epatite cronica non-A e non-B |
| Virus del vaiolo | DNA a doppio filamento | Vaiolo |
| Virus dell’epatite A | RNA a singolo filamento | Epatite acuta infettiva |
| Rinovirus | RNA a singolo filamento | Raffreddore comune |
| Virus dell’HIV | RNA a filamento singolo (retrovirus) | Sindrome da immunodeficienza acquisita (AIDS) |
| Virus della febbre gialla | RNA a filamento singolo | Febbre gialla |
Principali virus umani e patologie correlate
Diversi virus sono responsabili di malattie acute e croniche nell’essere umano. Essi presentano genomi a DNA o RNA, a singolo o doppio filamento, che determinano differenti strategie replicative. Le patologie associate spaziano dalle infezioni respiratorie comuni alle epatiti virali, fino a malattie sistemiche come l’AIDS e il vaiolo.
Tra i virus che infettano eucarioti, i retrovirus occupano una posizione peculiare poiché replicano l’informazione genetica in direzione inversa rispetto al percorso canonico DNA → RNA → proteina. In comune con i retrotrasposoni, essi utilizzano un’enzima chiave, la trascrittasi inversa, per sintetizzare DNA a partire da RNA. Una visione evolutiva largamente accettata ipotizza che i retrovirus siano derivati da antichi retrotrasposoni che, acquisendo geni per le proteine dell’involucro e per l’assemblaggio di particelle, hanno guadagnato la capacità di lasciare la cellula e diffondersi come virioni.
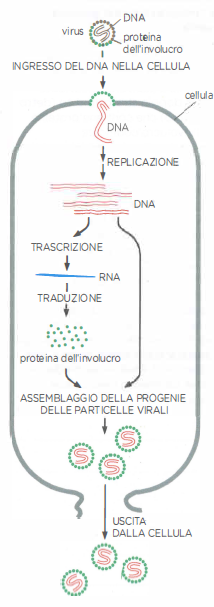
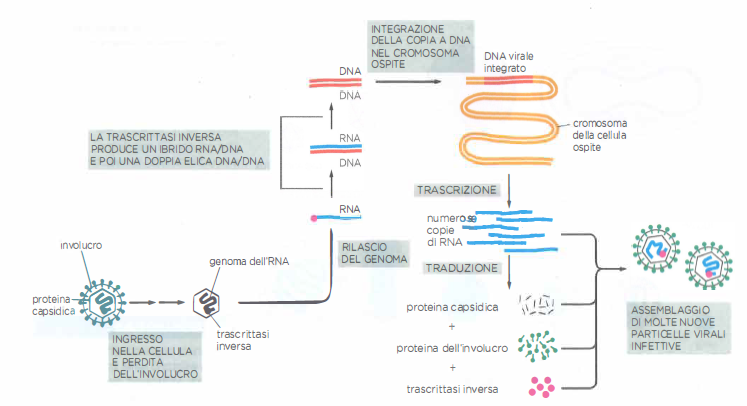
Il genoma retrovirale, costituito da RNA a singolo filamento, entra nella cellula insieme a molecole di trascrittasi inversa e ad altre proteine virali. La sintesi del DNA procede per stadi: l’enzima, spesso innescato da un tRNA cellulare, produce un ibrido DNA/RNA; l’attività RNasi H degrada l’RNA stampo; quindi viene sintetizzato il secondo filamento di DNA, ottenendo una doppia elica provirale dotata di LTR alle estremità. Un’altra proteina virale, l’integrasi, catalizza l’inserimento di questo DNA in una posizione del genoma dell’ospite selezionata con scarsa specificità di sequenza. L’integrazione genera così un provirus stabile, che viene copiato insieme al DNA cellulare a ogni divisione.
In condizioni favorevoli, il DNA provirale è trascritto dall’RNA polimerasi II dell’ospite, producendo RNA genomici e trascritti subgenomici per le proteine virali. Nella maggior parte dei retrovirus, il genoma codifica almeno le regioni gag (proteine del capside), pol (trascrittasi inversa, integrasi e proteasi) ed env (glicoproteine dell’involucro). L’assemblaggio avviene alla membrana plasmatica o in compartimenti intracellulari, dove i componenti virali si organizzano in nuove particelle che gemmano portando con sé una porzione di membrana arricchita in proteine Env. I passaggi di integrazione e replicazione sono schematizzati in (Figura 04.04-07).
L’HIV (Human Immunodeficiency Virus), agente eziologico dell’AIDS, è un retrovirus che sfrutta la latenza provirale per persistere nei serbatoi cellulari. Questa caratteristica rende difficoltosa l’eradicazione dell’infezione con i farmaci. La farmacoterapia ha tuttavia identificato bersagli esclusivamente virali o assenti nei processi cellulari essenziali, come la trascrittasi inversa; a questi si affiancano inibitori dell’integrasi e della proteasi, che, in combinazione, riducono drasticamente la replicazione virale e l’emergere di resistenze:
- Replica “al contrario”: sintesi di DNA da RNA tramite trascrittasi inversa e integrazione stabile nel genoma dell’ospite;
- Persistenza: stato provirale trasmesso alle cellule figlie finché segnali intracellulari o extracellulari non riattivano l’espressione virale;
- Strategie terapeutiche: inibizione enzimatica mirata (trascrittasi inversa, integrasi, proteasi) per bloccare tappe chiave del ciclo replicativo.