Le molteplici varietà strutturali delle proteine determinano l’ampia gamma delle loro funzioni
La modulazione dell’architettura cromosomica
Dopo aver descritto come il DNA venga organizzato nella cromatina, è necessario comprendere i meccanismi che permettono di modulare in modo dinamico tale impacchettamento per rendere il materiale genetico prontamente accessibile. Poiché il genoma eucariotico custodisce una quantità ingente di informazione, la cellula deve attivare strategie selettive che liberino segmenti precisi di DNA da una condizione compatta, così da consentire l’interazione con proteine regolatrici e complessi multiproteici deputati all’espressione genica, alla replicazione e alla riparazione.
Variazioni locali dei nucleosomi e accessibilità del DNA
Le cellule eucariotiche regolano in modo puntuale e rapido la disposizione dei nucleosomi per modulare l’accesso al DNA. Una via fondamentale implica l’azione di complessi rimodellatori della cromatina, apparati ATP-dipendenti che alterano la posizione e/o l’avvolgimento del DNA intorno all’ottamero istonico, promuovendo scorrimento, espulsione o inserimento di nucleosomi (Figura 02.04-01). Tali complessi, che riconoscono contemporaneamente DNA e istoni, riorganizzano la mappa nucleosomica per incrementare o ridurre la disponibilità di siti per fattori di trascrizione, polimerasi e altre proteine nucleari. Durante la mitosi molti rimodellatori risultano inibiti, circostanza che contribuisce al mantenimento della massima compattazione dei cromosomi.
Un secondo asse regolativo è costituito dalle modifiche covalenti e reversibili delle code istoniche, introdotte e rimosse da famiglie enzimatiche specializzate. Le code di H2A, H2B, H3 e H4 sono bersagli di acetilazione, metilazione, fosforilazione, ubiquitinazione e altre modifiche (Figura 02.04-02). Tali segnali influenzano sia le interazioni tra nucleosomi sia il reclutamento di proteine effettrici. Ad esempio, l’acetilazione delle lisine neutralizza cariche positive, attenua l’attrazione elettrostatica tra code e DNA e tra nucleosomi adiacenti, e determina un allentamento della fibra cromatinica che facilita il legame di fattori regolatori. Al contrario, specifiche metilazioni possono stabilizzare stati compatti o, in altri contesti, favorire l’attivazione, a seconda del residuo e del grado di metilazione.
Le modifiche istoniche operano anche come piattaforme di riconoscimento per domini proteici dedicati, dando luogo a circuiti di “scrittori” (writer), “lettori” (reader) e “cancellatori” (eraser). Schemi distinti di marcature reclutano complessi con funzioni diverse: alcuni promuovono condensazione, altri estensione della cromatina, altri ancora segnalano eventi quali replicazione recente o necessità di repressione/attivazione trascrizionale in segmenti specifici (Figura 02.04-02). In tal modo, combinazioni di segni istonici codificano istruzioni contestuali sull’uso locale dell’informazione genomica.
L’azione delle metiltransferasi, acetiltransferasi, deacetilasi, demetilasi e chinasi è strettamente indirizzata. Il targeting avviene per interazione con proteine leganti sequenze di DNA, con RNA non codificanti prodotti dal locus o con complessi già associati alla cromatina. La cooperazione tra rimodellatori ATP-dipendenti e modificatori degli istoni permette alla cellula di riconfigurare rapidamente porzioni di cromatina in risposta a stimoli e necessità fisiologiche.
Oltre alle modifiche delle code, concorrono alla plasticità cromatinica:
- l’incorporazione di varianti istoniche, come H3.3, H2A.Z e macroH2A, che alterano le proprietà del nucleosoma e segnalano stati funzionali distinti;
- l’azione di chaperoni istonici (ad esempio CAF-1, HIRA), che assistono l’assemblaggio/disassemblaggio nucleosomico durante trascrizione e replicazione;
- l’energia fornita dall’idrolisi di ATP, \(\Delta G_{ATP}\), che alimenta il rimodellamento meccanico del DNA avvolto sugli istoni.
Image Gallery

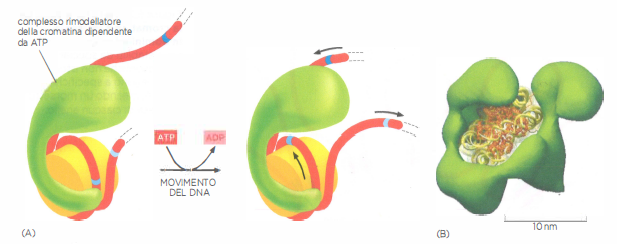
Complessi rimodellatori della cromatina
I complessi rimodellatori della cromatina riposizionano il DNA avvolto sul nucleosoma. (A) I complessi utilizzano l’energia derivante dall’idrolisi dell’ATP per distendere il DNA nucleosomico e farlo scivolare lungo l’ottamero istonico, esponendo il DNA ad altre proteine che legano il DNA. Le bande blu sono state aggiunte per mettere in evidenza lo spostamento del nucleosoma lungo il DNA. Uno slittamento di questo tipo richiede molti cicli di idrolisi dell’ATP. (B) Struttura di un complesso rimodellatore della cromatina, che mostra come l’enzima ospiti una particella nucleosomica, incluso un ottamero istonico (in arancione) e il DNA avvolto attorno ad esso (in verde chiaro). Questo grande complesso, purificato dal lievito, contiene 15 subunità, di cui una che idrolizza l’ATP e quattro che riconoscono specifici istoni modificati covalentemente.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery

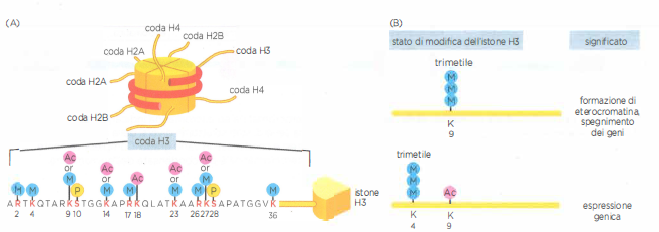
Modifiche delle code istoniche
Il quadro di modifiche delle code istoniche può determinare come la cellula tratterà un certo segmento di cromatina. (A) Rappresentazione schematica in cui viene mostrata la posizione delle code istoniche che si estendono da ogni nucleosoma. Ogni istone ha un certo numero di gruppi chimici che possono modificarlo, prevalentemente alle estremità, formando un legame covalente. All’estremità dell’istone H3, per esempio, può associarsi un gruppo acetilico (in sigla Ac), un gruppo metilico (M) o un gruppo fosfato (P). I numeri nella figura indicano la posizione dell’amminoacido modificato nella coda istonica, indicato dal suo identificativo in lettere. Si noti che alcune posizioni, per esempio le lisine (K) 9, 14, 23 e 27, possono essere modificate tramite acetilazione o metilazione (ma non da entrambe contemporaneamente). Inoltre, le lisine possono essere modificate con uno, due o tre gruppi metilici; per esempio, in (B) è rappresentata una trimetilazione. Si noti che l’istone H3 consta di 135 amminoacidi, per la maggior parte compresi nella zona globulare (rappresentata dal cuneo), e che la maggior parte delle modifiche interessa la sua coda N-terminale, di cui sono mostrati 36 amminoacidi. (B) Diverse combinazioni di modifiche delle code istoniche possono conferire un significato preciso al tratto di cromatina che li contiene, come indicato. Per il momento solo qualcuno dei “significati” di questo codice è stato chiarito.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery


Modifiche delle code istoniche
Il quadro di modifiche delle code istoniche può determinare come la cellula tratterà un certo segmento di cromatina. (A) Rappresentazione schematica in cui viene mostrata la posizione delle code istoniche che si estendono da ogni nucleosoma. Ogni istone ha un certo numero di gruppi chimici che possono modificarlo, prevalentemente alle estremità, formando un legame covalente. All’estremità dell’istone H3, per esempio, può associarsi un gruppo acetilico (in sigla Ac), un gruppo metilico (M) o un gruppo fosfato (P). I numeri nella figura indicano la posizione dell’amminoacido modificato nella coda istonica, indicato dal suo identificativo in lettere. Si noti che alcune posizioni, per esempio le lisine (K) 9, 14, 23 e 27, possono essere modificate tramite acetilazione o metilazione (ma non da entrambe contemporaneamente). Inoltre, le lisine possono essere modificate con uno, due o tre gruppi metilici; per esempio, in (B) è rappresentata una trimetilazione. Si noti che l’istone H3 consta di 135 amminoacidi, per la maggior parte compresi nella zona globulare (rappresentata dal cuneo), e che la maggior parte delle modifiche interessa la sua coda N-terminale, di cui sono mostrati 36 amminoacidi. (B) Diverse combinazioni di modifiche delle code istoniche possono conferire un significato preciso al tratto di cromatina che li contiene, come indicato. Per il momento solo qualcuno dei “significati” di questo codice è stato chiarito.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Nell’interfase la cromatina esibisce livelli multipli di compattazione
Le modulazioni locali della cromatina si riflettono sull’organizzazione dei cromosomi in interfase, che non presentano un unico stato strutturale. Le regioni che ospitano geni attivamente trascritti mostrano conformazioni relativamente aperte e dinamiche, mentre le zone con geni silenti tendono a essere più compatte. Ne consegue che il profilo di compattazione di un cromosoma varia tra tipi cellulari differenti, in accordo con l’assetto di espressione genica specifico. Nella maggior parte dei contesti cellulari, solo una frazione dei geni risulta espressa a livelli apprezzabili.
La forma più compatta di cromatina interfasica è l’eterocromatina, individuata storicamente come porzioni intensamente colorabili al microscopio ottico. Nei mammiferi, essa rappresenta approssimativamente il 10% della cromatina in interfase e si accumula soprattutto in prossimità di centromeri e telomeri (Figura 02.04-07). La rimanente parte è definita eucromatina, più accessibile in media, benché sia oggi evidente che sia eterocromatina sia eucromatina corrispondano in realtà a insiemi compositi di stati cromatinici con gradi intermedi di compattazione (Figura 02.04-03).
Specifiche combinazioni di modifiche istoniche stabilizzano e mantengono questi stati. Per l’eterocromatina costitutiva, una marcatura cardine è la trimetilazione della lisina 9 dell’istone H3 (H3K9me3) (Figura 02.04-02). Una volta instaurato, questo segnale viene riconosciuto da proteine leganti metilisina (per esempio HP1), che reclutano a loro volta ulteriori “writer” capaci di propagare H3K9me3 ai nucleosomi vicini. Il risultato è un’onda di compattazione che può estendersi lungo il cromosoma finché non incontra sequenze barriera che interrompono la diffusione (Figura 02.04-04).
Le barriere possono agire in svariati modi:
- contenere siti per fattori che reclutano acetiltransferasi, capaci di acetilare H3K9 e impedire la sua metilazione successiva;
- ospitare proteine isolatrici e architetturali, come CTCF con cofattori, che organizzano confini e domini topologici;
- produrre RNA non codificanti che interferiscono con il reclutamento dei complessi di propagazione dell’eterocromatina.
In genere, i tratti stabilmente eterocromatici sono poveri di geni. Quando un gene funzionale viene inglobato in eterocromatina per effetto della posizione, la sua espressione diviene inefficiente o variabile (fenomeno analogo alla variegazione da effetto posizione descritta in diversi modelli animali). La deregolazione derivante da spostamenti dei confini cromatinici può contribuire a patologie: ad esempio, la contrazione o l’espansione di regioni ripetitive che rimodellano l’assetto eterocromatinico è implicata in disordini ereditari del tessuto muscolare e del neurosviluppo, a testimonianza della sensibilità dei geni al contesto cromatinico circostante.
Un caso paradigmatico di silenziamento cromatinico stabile su vasta scala è l’inattivazione del cromosoma X nelle femmine dei mammiferi. Poiché le cellule femminili possiedono due cromosomi X e quelle maschili un X e un Y, è necessario un meccanismo di compensazione del dosaggio: in ciascuna cellula femminile, in fase precoce dello sviluppo embrionale, uno dei due cromosomi X viene selezionato in modo stocastico e convertito in un’unità eterocromatinica in gran parte trascrizionalmente inattiva, nota anche come corpo di Barr. Questo stato si mantiene nelle linee cellulari discendenti (Figura 02.04-05). Il fenomeno produce mosaici di espressione genica X-linked, visibili anche a livello fenotipico in vari organismi; un esempio classico è la pigmentazione a chiazze dei gatti calico, dovuta all’inattivazione differenziale dell’X nei melanociti (Figura 02.04-06). A livello molecolare, l’RNA lungo non codificante Xist riveste il cromosoma destinato all’inattivazione e guida il reclutamento di complessi repressivi e di modificatori istonici, tra cui marcature come H3K27me3 e macroH2A, che contribuiscono alla compattazione e al silenziamento.
Processi analoghi, anche se più circoscritti, intervengono diffusamente per definire profili di espressione genica cellula-specifici. All’atto della divisione, le cellule trasmettono con alta fedeltà non solo la sequenza del DNA, ma anche aspetti della sua organizzazione: le varianti istoniche, porzioni delle marcature post-traduzionali e configurazioni tridimensionali del cromosoma vengono ripristinate dopo la replicazione grazie a chaperoni e complessi “writer/reader” che copiano i pattern preesistenti su nucleosomi neoassemblati. Tale memoria epigenetica, comprendente anche la metilazione del DNA laddove presente, codifica quali geni rimangono attivi o repressi e sostiene la stabilità dell’identità cellulare nei tessuti di un organismo pluricellulare complesso.
In sintesi, l’accesso al DNA è controllato dall’integrazione di più livelli:
- rimodellamento meccanico dei nucleosomi, che riposiziona o rimuove gli istoni per scoprire o coprire sequenze regolatrici;
- modifiche istoniche, che indeboliscono o rafforzano le interazioni cromatiniche e fungono da codici di reclutamento di complessi effettrici;
- barriere e domini cromosomici che delimitano la propagazione degli stati compatti o aperti;
- incorporazione di varianti istoniche e azione di RNA non codificanti, che stabilizzano stati funzionali;
- meccanismi di memoria che replicano i pattern epigenetici durante il ciclo cellulare.
Questi principi, consolidati da studi classici e contemporanei in biologia molecolare e cellulare, spiegano come la cromatina possa simultaneamente proteggere il genoma, comprimerlo in strutture maneggevoli e, quando richiesto, esporre rapidamente l’informazione necessaria per l’esecuzione dei programmi cellulari.
Image Gallery

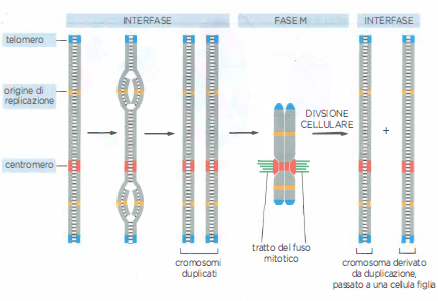
Elementi essenziali del cromosoma
Tre sono gli elementi che devono essere presenti nella sequenza del DNA perché il cromosoma eucariote possa replicarsi e poi segregare al momento della mitosi. Ogni cromosoma ha origini di replicazione multiple, un centromero e due telomeri. Nella figura è schematizzata la serie di eventi che interessano un cromosoma tipico durante il ciclo cellulare. Durante l’interfase il DNA si replica a partire dalle origini di replicazione in entrambe le direzioni e per tutta la sua lunghezza. Nella fase M il centromero attacca ognuno dei cromosomi duplicati al fuso mitotico, in modo che, quando la cellula si divide, una copia venga distribuita a ognuna delle cellule figlie. Prima della divisione cellulare, il centromero serve anche a mantenere insieme i cromosomi duplicati finché non sono pronti a separarsi. I telomeri contengono sequenze di DNA che consentono la duplicazione completa delle estremità cromosomiche.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery

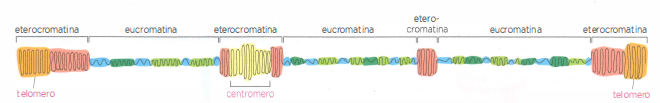
Struttura della cromatina
La struttura della cromatina varia lungo il cromosoma interfascico. Come schematicamente indicato dai diversi colori (e dal percorso delle molecole di DNA rappresentato da una linea nera centrale), i termini eterocromatina ed eucromatina indicano ciascuno una serie di stati strutturali diversi della cromatina, estesi e condensati in vario grado. Complessivamente l’eterocromatina è più condensata dell’eucromatina.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery


Modifiche delle code istoniche
Il quadro di modifiche delle code istoniche può determinare come la cellula tratterà un certo segmento di cromatina. (A) Rappresentazione schematica in cui viene mostrata la posizione delle code istoniche che si estendono da ogni nucleosoma. Ogni istone ha un certo numero di gruppi chimici che possono modificarlo, prevalentemente alle estremità, formando un legame covalente. All’estremità dell’istone H3, per esempio, può associarsi un gruppo acetilico (in sigla Ac), un gruppo metilico (M) o un gruppo fosfato (P). I numeri nella figura indicano la posizione dell’amminoacido modificato nella coda istonica, indicato dal suo identificativo in lettere. Si noti che alcune posizioni, per esempio le lisine (K) 9, 14, 23 e 27, possono essere modificate tramite acetilazione o metilazione (ma non da entrambe contemporaneamente). Inoltre, le lisine possono essere modificate con uno, due o tre gruppi metilici; per esempio, in (B) è rappresentata una trimetilazione. Si noti che l’istone H3 consta di 135 amminoacidi, per la maggior parte compresi nella zona globulare (rappresentata dal cuneo), e che la maggior parte delle modifiche interessa la sua coda N-terminale, di cui sono mostrati 36 amminoacidi. (B) Diverse combinazioni di modifiche delle code istoniche possono conferire un significato preciso al tratto di cromatina che li contiene, come indicato. Per il momento solo qualcuno dei “significati” di questo codice è stato chiarito.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery

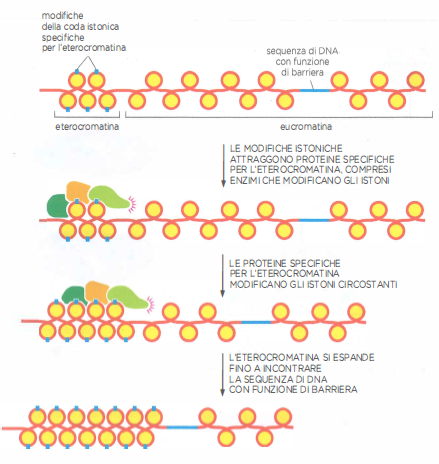
Formazione dell’eterocromatina
Le modifiche specifiche per l’eterocromatina permettono la formazione e l’estensione dell’eterocromatina. Queste modifiche attraggono proteine specifiche per l’eterocromatina che riproducono le stesse modifiche sui nucleosomi circostanti. In questo modo, l’eterocromatina può espandersi fino a quando incontra la sequenza di DNA che funge da barriera, bloccando la sua propagazione nelle regioni di eucromatina.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery

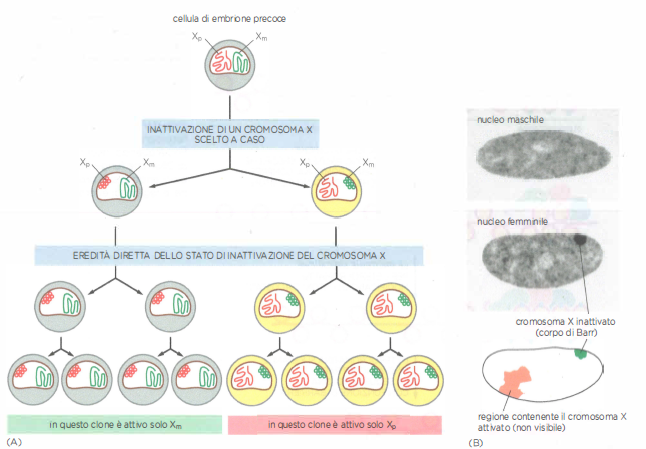
Inattivazione del cromosoma X
Uno dei due cromosomi X può venire inattivato nelle cellule di mammifero femmina per formazione di eterocromatina. (A) Negli individui femmina, ogni cellula contiene due cromosomi X, uno proveniente dalla madre (Xᵐ) e l’altro dal padre (Xᵖ). In ogni cellula uno di questi due cromosomi si condensa in eterocromatina, già allo stadio iniziale dello sviluppo, apparentemente a caso. Lo stesso cromosoma si condensa (s’inattiva) da questo evento in poi a ogni divisione cellulare e in tutti i discendenti di quella cellula. Perciò, tutte le femmine di mammifero diventano un mosaico di cellule che portano come cromosoma X inattivato quello materno o quello paterno: quasi tutti i loro tessuti e organi avranno circa una metà di cellule di un tipo e una metà dell’altro. (B) Nel nucleo di una cellula femminile, il cromosoma X inattivato può essere visto come una piccola e distinta massa di cromatina chiamata corpo di Barr, dal nome del medico che per primo lo ha osservato. In queste microfotografie di nuclei di fibroblasti umani, il cromosoma X inattivato nel nucleo femminile (microfotografia inferiore) è stato visualizzato mediante l’uso di un anticorpo che riconosce le proteine associate al corpo di Barr. Il nucleo maschile (in alto) contiene un unico cromosoma X, che non è inattivato e quindi non è riconosciuto da questo anticorpo. Sotto le microfotografie, un’illustrazione mostra le posizioni dei cromosomi X sia attivi sia inattivi nel nucleo femminile.
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.
Image Gallery


Colore del mantello nei gatti calico
Il colore del mantello di un gatto calico dipende in gran parte dall’inattivazione dell’X. Nei gatti, uno dei geni che specifica il colore del mantello si trova sul cromosoma X. Nei calico femmine, un cromosoma X contiene il gene che specifica il pelo nero, l’altro contiene il gene che specifica il pelo arancione. Le cellule cutanee in cui è inattivato il cromosoma X che contiene il gene per il pelo nero producono peli arancioni; quelle in cui è inattivato il cromosoma X che contiene il gene per il pelo arancione producono peli neri. La dimensione di ogni zona dipende dal numero di cellule cutanee che sono discese da una cellula embrionale in cui uno o l’altro dei cromosomi X è stato inattivato in modo casuale durante lo sviluppo (Figura 02.04-05).
Immagine tratta liberamente da Internet. Se viola i tuoi diritti, contattaci.















