Il metabolismo di un organismo trasforma materia ed energia
Definizione
L’espressione genica è l’insieme coordinato di processi mediante i quali le cellule attivano in modo selettivo la produzione di RNA e proteine a partire dall’informazione contenuta nel genoma. Attraverso reti regolatorie di fattori di trascrizione, cromatina e RNA non codificanti, una singola cellula decide quali geni debbano essere accesi o spenti, con quale intensità e in quale momento. Tale selettività è cruciale durante lo sviluppo embrionale e nei tessuti adulti, perché consente la progressiva specializzazione in cellule muscolari, neuroni, cellule ematiche e numerosi altri tipi cellulari. L’identità cellulare deriva, in ultima analisi, dall’accumulo di specifici insiemi di trascritti e proteine, cioè da programmi di espressione genica distinti che definiscono uno specifico trascrittoma e proteoma.
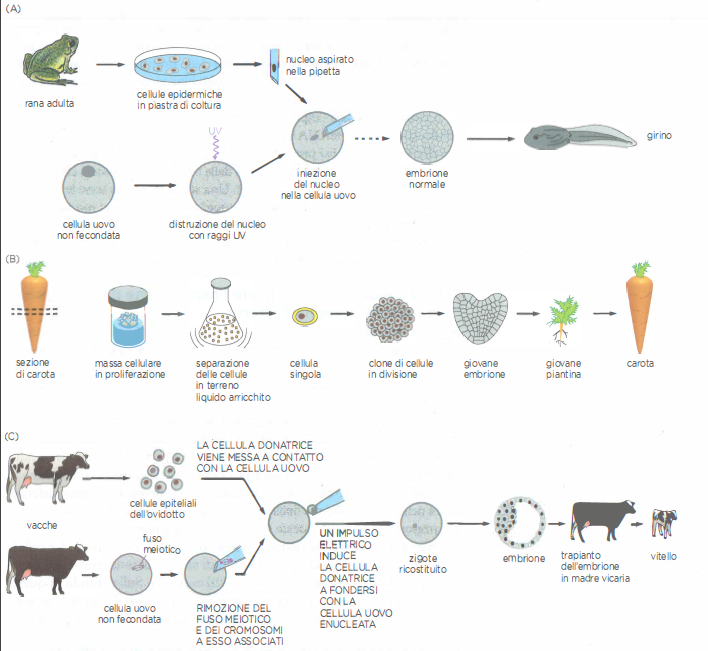
La differenziazione cellulare non implica una riscrittura irreversibile della sequenza del DNA nucleare. Ciò è stato dimostrato da esperimenti di trapianto nucleare: il nucleo di una cellula somatica differenziata, trasferito in un ovocita enucleato, può sostenere lo sviluppo completo di un organismo. Ad esempio, il nucleo di una cellula epidermica di rana, iniettato in una cellula uovo priva di nucleo, è in grado in alcuni casi di sostenere lo sviluppo fino al girino (Figura 03.09-01), indicando che non sono state perse informazioni genetiche essenziali. Approcci analoghi sono stati applicati ai mammiferi, portando alla generazione di organismi a partire da cellule differenziate di pecora, bovino, maiale, capra e topo. Nelle piante, la totipotenza è particolarmente evidente: cellule isolate di carota, ad esempio, possono rigenerare un intero individuo adulto.
Queste osservazioni supportano il principio di equivalenza nucleare: i tipi cellulari specializzati mantengono, in linea generale, lo stesso repertorio di geni. Le differenze funzionali tra cellule dipendono quindi da schemi regolatori di espressione e da modifiche epigenetiche ereditabili (come metilazione del DNA e modificazioni istoniche) che influenzano l’accessibilità ai geni senza cambiare la sequenza nucleotidica. Fanno eccezione casi specifici e localizzati, come il riarrangiamento somatico dei loci delle immunoglobuline nei linfociti o variazioni del DNA mitocondriale; tali eccezioni, tuttavia, non inficiano il principio generale.
La misura delle differenze tra tipi cellulari può essere ottenuta confrontando i rispettivi proteomi. Storicamente si impiegava l’elettroforesi bidimensionale su gel per separare le proteine; oggi la spettrometria di massa accoppiata a cromatografia liquida (LC–MS/MS), in modalità dipendente o indipendente dai dati, consente identificazioni più sensibili e quantificazioni più accurate, includendo proteine a bassa abbondanza e diverse proteoforme. Entrambi gli approcci mostrano che un’ampia frazione del proteoma è condivisa tra i tipi cellulari e comprende proteine di “manutenzione” (housekeeping) necessarie alla sopravvivenza cellulare:
- polimerasi dell’RNA e fattori accessori essenziali per la trascrizione;
- proteine coinvolte nella riparazione del DNA e nel controllo del ciclo cellulare;
- componenti ribosomiali e fattori di traduzione;
- enzimi centrali del metabolismo, come quelli della glicolisi e del ciclo degli acidi tricarbossilici;
- elementi del citoscheletro, tra cui actina, tubulina e proteine dei filamenti intermedi.
Accanto a questo nucleo comune, ciascun tipo cellulare produce proteine specializzate che ne determinano i tratti distintivi; per esempio, i globuli rossi in maturazione sintetizzano principalmente emoglobina, mentre i neuroni esprimono canali ionici e enzimi per la sintesi di neurotrasmettitori.
Un quadro complementare deriva dall’analisi del trascrittoma. Le tecniche di sequenziamento dell’RNA (RNA‑seq), incluse varianti a singola cellula, permettono di catalogare l’insieme degli RNA, mRNA compresi, e di stimarne l’abbondanza relativa. Nelle cellule umane differenziate, in un dato momento, risultano tipicamente attivi circa 6 000–12 000 geni codificanti proteine su un totale di circa \( \sim 19\,000 \) geni codificanti; il numero preciso dipende dal tipo cellulare e dallo stato fisiologico. Le analisi rivelano anche la presenza estesa di isoforme generate da splicing alternativo e l’abbondanza di RNA non codificanti (rRNA, tRNA, snRNA, miRNA e lncRNA), con ruoli strutturali e regolatori nel controllo dell’espressione.
I programmi di espressione, pur stabili nel definire l’identità cellulare, restano dinamici e rispondono a ormoni, fattori di crescita, nutrienti e segnali paracrini. Nelle cellule epatiche, ad esempio, l’ormone steroideo cortisolo si lega al recettore glucocorticoide, che agisce come fattore di trascrizione e rimodella l’attività di interi insiemi di geni. L’effetto include l’aumento della produzione di glucosio a partire da amminoacidi e altre piccole molecole, con induzione coordinata di enzimi chiave, tra cui la tirosina aminotransferasi, la cui sintesi cresce sensibilmente durante il segnale e torna ai livelli basali quando l’ormone viene rimosso. Altri tipi cellulari mostrano risposte differenti: negli adipociti, per esempio, la stessa via può sopprimere la sintesi di specifici enzimi, mentre alcune cellule restano poco responsive. Questa diversità dipende dal repertorio di fattori di trascrizione disponibili, dallo stato della cromatina e dalla presenza di elementi regolativi (enhancer) accessibili in ciascun tipo cellulare.
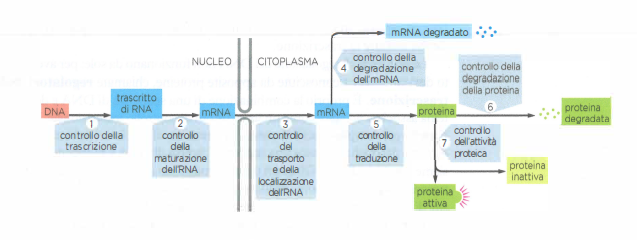
Poiché il flusso dell’informazione genetica procede attraverso numerosi passaggi, in linea di principio ogni fase offre un punto di controllo (Figura 03.09-02). La cellula può modulare la produzione proteica con strategie complementari:
- determinando l’avvio e la frequenza della trascrizione di ciascun gene, attraverso cis‑elementi (promotori, enhancer) e proteine regolatrici che agiscono sullo stato della cromatina e sul reclutamento della RNA polimerasi;
- rimodellando i trascritti primari mediante splicing alternativo, scelta del sito di poliadenilazione, editing dell’RNA e modifiche delle code, per generare isoforme con proprietà diverse;
- selezionando quali mRNA esportare dal nucleo al citoplasma e in quali tempi, tramite complessi di legame all’mRNA e segnali di localizzazione;
- controllando la stabilità degli mRNA, attraverso sequenze regolative nelle UTR e fattori che ne determinano la degradazione o il silenziamento mediato da miRNA;
- regolando l’efficienza di traduzione sui ribosomi, per esempio tramite la fosforilazione di fattori di inizio e l’organizzazione in granuli citoplasmatici;
- modulando la durata di vita delle proteine neo-sintetizzate, con modifiche post‑traduzionali (es. ubiquitinazione) che ne indirizzano la degradazione nel proteasoma o nei lisosomi.
A valle della sintesi, l’attività proteica può essere ulteriormente regolata mediante fosforilazione, acetilazione, legame di ligandi, cambiamenti conformazionali e compartimentalizzazione subcellulare, assicurando una risposta rapida agli stimoli. Tra tutti i livelli, il controllo trascrizionale è spesso predominante perché evita la produzione di intermedi non necessari e consente una regolazione ampia e coordinata di interi moduli genici.