Le vie cataboliche liberano energia ossidando i combustibili organici
Definizione
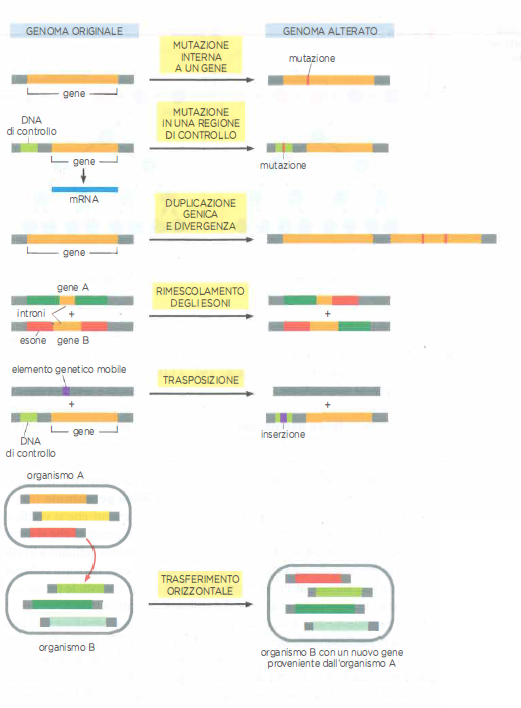
L’innovazione genetica, nella maggior parte dei casi, non scaturisce dalla comparsa ex novo di lunghi tratti di DNA, ma dall’elaborazione di materiale preesistente. L’evoluzione agisce soprattutto riconfigurando, combinando e modulando sequenze ereditate dagli antenati, generando un ventaglio di forme e funzioni a partire da “motivi” molecolari già presenti nelle popolazioni. In rari casi si osserva l’origine de novo di geni da regioni non codificanti, ma i contributi prevalenti alla diversità biologica derivano dalla rielaborazione di sequenze ancestrali. L’accumulo, lungo milioni di generazioni, di piccole e grandi modificazioni può condurre a trasformazioni profonde. Gran parte di questi cambiamenti ricade in alcune classi generali (Figura 04.02-01):
- Mutazioni intrageniche: un gene esistente può essere alterato da sostituzioni di singoli nucleotidi, da inserzioni o delezioni di varia estensione oppure da micro-riarrangiamenti locali; tali eventi possono modificare lo splicing, influenzare la stabilità dell’RNA, alterare la struttura o la localizzazione subcellulare della proteina, o incidere sulle sue interazioni molecolari;
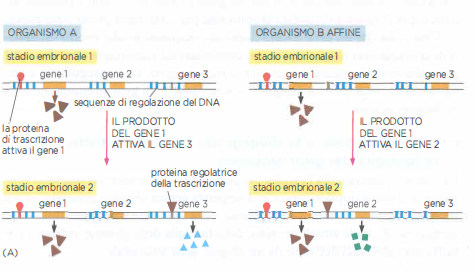
- Variazioni nelle sequenze regolative: mutazioni in promotori, enhancer, silencer o insulator possono ridefinire tempi, livelli e sedi di espressione di un gene, con effetti fenotipici spesso rilevanti; molte differenze tra linee evolutive distanti, come pesci e umani, riflettono divergenze nella regolazione trascrizionale più che nel repertorio genico;
- Duplicazione e divergenza: copie aggiuntive di geni, segmenti cromosomici o interi genomi possono fissarsi e poi divergere attraverso mutazione e selezione, portando a neofunzionalizzazione, subfunzionalizzazione o aggiustamenti di dosaggio; tali duplicazioni avvengono su scale che vanno dalla duplicazione tandem alle poliploidizzazioni;
- Rimescolamento di esoni (exon shuffling): ricombinazioni che avvengono preferenzialmente negli introni possono combinare domini proteici provenienti da geni diversi, generando configurazioni modulari nuove; la compatibilità di fase degli introni facilita il mantenimento della cornice di lettura senza necessità di giunzioni perfettamente precise;
- Trasposizione di elementi mobili: retrotrasposoni e trasposoni a DNA possono inserirsi vicino o dentro i geni, modificandone l’attività o fungendo da serbatoi di sequenze regolative; essi promuovono anche duplicazioni, delezioni e riarrangiamenti, contribuendo in modo sostanziale alla plasticità del genoma;
- Trasferimento orizzontale di geni: l’acquisizione di DNA da organismi non parentali, frequente nei batteri tramite trasformazione, trasduzione o coniugazione, e rara ma documentata negli eucarioti, introduce tratti genetici completi al di fuori della discendenza verticale.
Ognuna di queste sorgenti di variazione ha contribuito all’evoluzione dei genomi attuali e continua a operare. La trasmissione del cambiamento, tuttavia, dipende dal contesto riproduttivo: nelle specie a fecondazione sessuata soltanto una parte delle cellule può veicolare le novità alle generazioni successive. Prima di analizzare in dettaglio i meccanismi di modifica del DNA e le loro conseguenze evolutive, è opportuno chiarire come la riproduzione sessuale condizioni l’ereditarietà delle varianti.
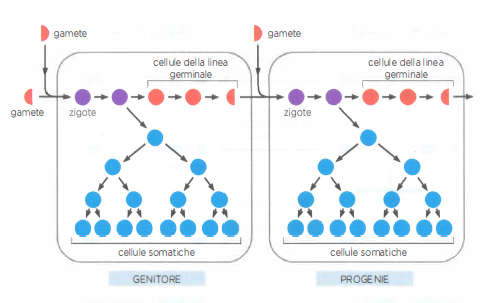
Negli organismi unicellulari che si riproducono per via asessuata, la genealogia coincide con la sequenza delle divisioni cellulari: ogni cellula replica il proprio genoma e lo distribuisce alla progenie. Nei metazoi e in molti altri organismi pluricellulari a riproduzione sessuata, invece, l’ereditarietà è vincolata alla linea germinale: soltanto i gameti trasmettono alle generazioni future una copia del genoma. Le cellule somatiche, pur cruciali per lo sviluppo e la sopravvivenza dell’organismo, non generano discendenti propri (Figura 04.02-02).
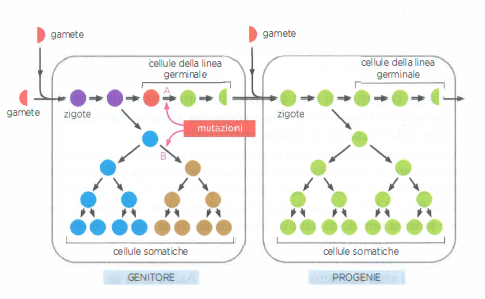
Ne consegue che una mutazione insorta in una cellula somatica, sebbene possa influire sulla salute dell’individuo, per esempio predisponendo all’insorgenza di neoplasie, non viene ereditata dalla sua prole. Perché una variante genetica contribuisca all’evoluzione di una popolazione, deve comparire in una cellula della linea germinale o in una cellula progenitrice dei gameti e raggiungere il gamete che parteciperà alla fecondazione (Figura 04.02-03). Questo principio, spesso indicato come “barriera di Weismann”, è particolarmente netto in molti animali; in altri gruppi, come numerose piante in cui la linea germinale si definisce tardivamente, possono verificarsi eccezioni locali, ma l’ereditarietà rimane legata ai gameti prodotti.
La riproduzione sessuata introduce inoltre una componente autonoma di variabilità genetica. Durante la meiosi, la segregazione indipendente dei cromosomi omologhi e il crossing-over generano combinazioni alleliche nuove a ogni generazione. Trascurando il ricombinamento, il numero di assetti cromosomici possibili è pari a \(2^n\), dove \(n\) è il numero di coppie di cromosomi; a questo si aggiunge la rimescolanza intra-cromosomica indotta dai chiasmi, che amplia ulteriormente lo spazio delle combinazioni. La fusione di gameti provenienti da due individui distinti unisce questi assortimenti in configurazioni genetiche uniche, facendo sì che la progenie differisca da entrambi i genitori.
Attraverso la successione ininterrotta delle divisioni nella linea germinale, ogni organismo sessuato è collegato ai suoi antenati e, risalendo nel tempo, alle prime cellule apparse oltre 3,5 miliardi di anni fa. In questo contesto, gli eventi mutazionali, i riarrangiamenti e le variazioni di numero di copie che insorgono nella linea germinale costituiscono la materia prima su cui agiscono selezione naturale, deriva genetica e migrazione, modellando nel lungo periodo l’architettura e la funzione dei genomi.
Le cellule eucariotiche e procariotiche dispongono di meccanismi estremamente accurati per duplicare e preservare l’informazione genetica, tra cui la correzione di bozze delle DNA polimerasi e i sistemi di riparo post-replicativo. Nonostante ciò, a ogni ciclo cellulare esiste una probabilità, pur se minima, che una singola coppia di basi venga alterata: si parla in questo caso di mutazioni puntiformi. Tali eventi derivano per lo più da errori sporadici di copiatura durante la replicazione o da riparazioni imperfette del DNA danneggiato.
Esperimenti condotti su colture di Escherichia coli hanno consentito di stimare direttamente il tasso di mutazione per nucleotide, pari a circa \( \mu \approx 3 \times 10^{-10} \) cambiamenti per coppia di basi per generazione cellulare. In condizioni di laboratorio, E. coli raddoppia ogni 20–25 minuti; in meno di 24 ore, una singola cellula originaria può generare una popolazione più numerosa dell’attuale popolazione umana. In una coltura di 109 cellule, ciò si traduce nella presenza di milioni di varianti genetiche indipendenti, ognuna portatrice di alterazioni quasi impercettibili rispetto al genoma della cellula progenitrice. Con un genoma di circa 4,6 milioni di coppie di basi (4,6 × 106), il numero atteso di mutazioni puntiformi nuove per genoma e per divisione è nell’ordine di \( 4{,}6 \times 10^{6} \times 3 \times 10^{-10} \approx 1{,}4 \times 10^{-3} \); ne consegue che, nella grande maggioranza dei casi (circa il 99,9% considerando un’approssimazione di Poisson), le due cellule figlie ereditano una sequenza identica a quella parentale, e solo raramente compaiono cellule mutanti.
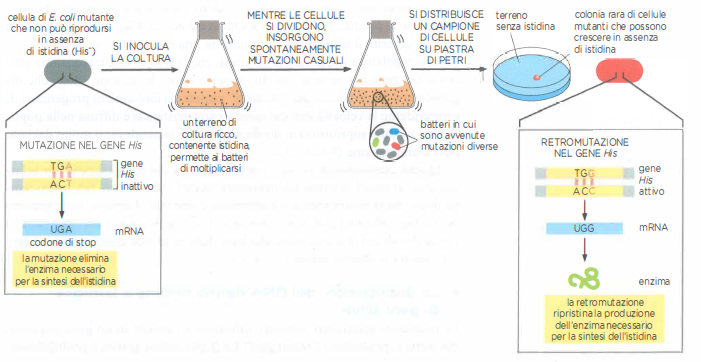
Le mutazioni puntiformi possono generare fenotipi selezionabili quando l’ambiente esercita una pressione specifica. L’aggiunta di un antibiotico, la presenza di un tossico o la privazione di un nutriente essenziale consentono di isolare, all’interno della popolazione, le cellule che hanno acquisito l’alterazione utile alla sopravvivenza: un approccio che permette di individuare il “mutante giusto” in mezzo a un’enorme maggioranza di cellule sensibili (Figura 04.02-04). Misure di questo tipo hanno corroborato la stima di una frequenza complessiva di circa 3 cambiamenti ogni 1010 coppie di basi per generazione in E. coli. Nei vertebrati, stime ottenute confrontando le sequenze genomiche di genitori e figli indicano per l’uomo un tasso per nucleotide di ordine comparabile, in media circa un terzo di quello osservato in E. coli, a testimonianza di una elevata efficienza dei meccanismi di fedeltà replicativa in specie evolutivamente distanti.
Gli esiti funzionali delle mutazioni puntiformi possono essere diversi:
- perdita di funzione di un gene, ad esempio attraverso una sostituzione che introduca un codone di stop prematuro o comprometta un sito catalitico;
- guadagno o aumento di funzione, evento raro ma possibile, per esempio tramite una sostituzione aminoacidica che migliori l’attività o la stabilità di una proteina (Figura 04.02-04);
- neutralità apparente, quando l’alterazione non produce cambiamenti fenotipici rilevabili.
Le mutazioni neutrali si riscontrano frequentemente in tratti genomici con vincoli funzionali deboli (per esempio alcune regioni introniche o intergeniche). Anche all’interno degli esoni possono risultare neutre se colpiscono la terza posizione di un codone senza modificare l’amminoacido codificato (mutazioni sinonime) o se determinano una sostituzione conservativa tra amminoacidi con proprietà biochimiche simili, con impatto minimo sulla struttura e funzione della proteina.
Non tutte le mutazioni puntiformi si collocano nelle regioni codificanti. Alterazioni di singoli nucleotidi in elementi regolatori del DNA, come promotori, enhancer o siti di legame per fattori trascrizionali, possono modificare la quantità, il luogo e il momento in cui un gene viene espresso. Poiché la regolazione dell’espressione è cruciale per l’omeostasi cellulare e l’adattamento, anche piccole variazioni in queste sequenze possono avere conseguenze fenotipiche rilevanti.
Un esempio classico riguarda la resistenza a Plasmodium vivax. Una mutazione puntiforme nel promotore del gene che codifica il recettore eritrocitario Duffy/ACKR1 riduce l’attività di un sito di legame per fattori trascrizionali eritroidi, impedendo l’espressione del recettore sulla superficie dei globuli rossi. Poiché P. vivax utilizza tale recettore per invadere l’eritrocita, gli individui portatori dell’allele inattivante nei globuli rossi risultano sostanzialmente refrattari all’infezione (protezione mediata dall’assenza del recettore bersaglio), un chiaro esempio di effetto regolativo con risvolti adattativi a livello di popolazione.
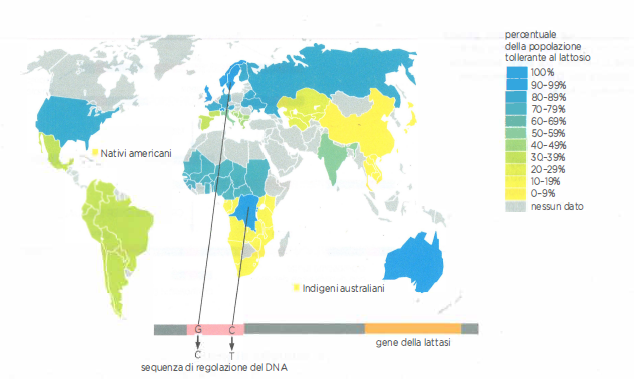
Un secondo caso emblematico è la persistenza della lattasi in età adulta. Nei gruppi umani antichi, l’enzima lattasi-florespina (LCT) veniva espresso prevalentemente durante l’infanzia; nelle popolazioni che, a partire da circa 10 000 anni fa, hanno praticato la pastorizia e consumato latte animale, si sono diffuse varianti regolative che mantengono elevata l’attività del gene LCT anche dopo lo svezzamento. Si tratta di mutazioni puntiformi localizzate in elementi enhancer all’interno del gene MCM6, che controllano a distanza la trascrizione di LCT. Tali varianti aumentano il reclutamento dei fattori trascrizionali nelle cellule dell’epitelio intestinale, sostenendo l’espressione della lattasi per tutta la vita. Dal punto di vista evolutivo, le persone adulte che digeriscono il lattosio rappresentano quindi “mutanti” regolativi rispetto ai loro antenati intolleranti, e la rapidità con cui questi alleli si sono diffusi in popolazioni ad alta dipendenza dai prodotti lattiero-caseari è notevole (Figura 04.02-05).
Mutazioni regolative analoghe, occorse in tempi sia recenti sia remoti, hanno contribuito a modellare differenze fenotipiche tra popolazioni e specie. Cambiamenti in sequenze di controllo che modulano l’intensità o il pattern spaziale e temporale dell’espressione genica possono produrre effetti morfologici e fisiologici profondi senza alterare le regioni codificanti delle proteine. Per questa ragione, la variabilità nelle sequenze regolatrici è considerata una delle principali fonti di innovazione evolutiva, oltre che una causa comune di fenotipi umani complessi e tratti adattativi (Figura 04.02-06).
Le mutazioni puntiformi modulano l’attività di geni già esistenti, ma la comparsa di nuovi geni deriva in gran parte dalla duplicazione genica. Quando un gene viene copiato, le due copie non sono uguali dal punto di vista evolutivo: in genere una rimane vincolata a mantenere la funzione originaria sotto selezione purificante, mentre l’altra può accumulare variazioni senza compromettere la sopravvivenza dell’organismo. Tali variazioni possono condurre, nel tempo, a nuove proprietà, secondo dinamiche di neofunzionalizzazione o, talvolta, di suddivisione delle funzioni ancestrali tra le copie (subfunzionalizzazione). Ripetuti cicli di duplicazione seguiti da divergenza, protratti per milioni di anni, trasformano un singolo gene in un’intera famiglia di geni “paraloghi”, ciascuno specializzato in compiti distinti all’interno dello stesso genoma. Le analisi comparative delle sequenze confermano l’ampiezza del fenomeno: in Bacillus subtilis, per esempio, una frazione consistente del genoma mostra paralogie interne ben riconoscibili.
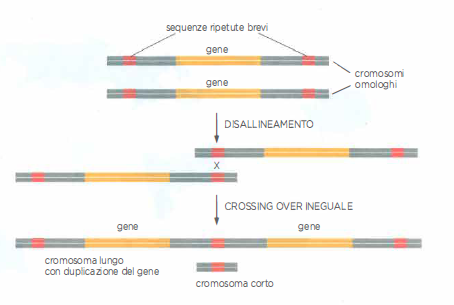
Come avvengono le duplicazioni? Un meccanismo chiave è la ricombinazione omologa, che in condizioni fisiologiche ripara rotture a doppio filamento usando una copia intatta come stampo. In particolare, allineamenti imperfetti tra brevi ripetizioni simili poste ai lati di un gene possono innescare crossing over non allelici (o “ineguali”), generando un cromosoma con una copia aggiuntiva e il cromosoma complementare privo del gene corrispondente (Figura 04.02-07). Il cromosoma accorciato tende a essere eliminato nei cicli successivi, mentre la copia duplicata può fungere da punto di partenza per ulteriori espansioni per lo stesso processo. Non sorprende, pertanto, che gruppi di geni fortemente affini si trovino spesso in serie in tandem.
Altri percorsi concorrono alla creazione di duplicati: duplicazioni segmentali dovute a eventi di ricombinazione tra ripetizioni a bassa copia, scivolamento della DNA polimerasi durante la replicazione, e retroposizione mediata da retrotrasposoni che produce copie prive di introni (retrogeni). Questi meccanismi, insieme, alimentano il serbatoio di varianti su cui agiscono selezione naturale e deriva genetica.
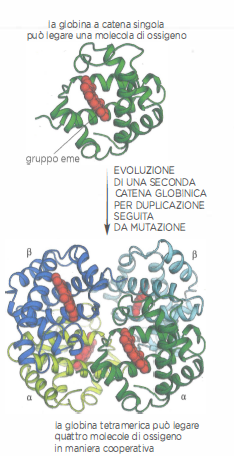
Le globine rappresentano un modello paradigmatico di come la duplicazione genica, seguita da modifica progressiva, generi nuove funzioni proteiche. Le omologie di sequenza e struttura tra le globine moderne indicano l’origine da un unico gene ancestrale. La forma più semplice è una proteina a catena singola di all’incirca 150 residui, presente in vari invertebrati e pesci basali, capace di legare e trasportare ossigeno.
Nei mammiferi e nella maggior parte dei vertebrati, la funzione di trasporto dell’ossigeno è affidata a un tetramero α₂β₂ (Figura 04.02-08). I quattro siti di legame cooperano allostericamente: il passaggio tra stati conformazionali a bassa e alta affinità consente un’associazione e un rilascio dell’ossigeno altamente efficienti, irraggiungibili per una globina monomerica. Questa cooperatività è cruciale per organismi di grandi dimensioni, in cui la diffusione semplice dell’ossigeno non garantisce un’ossigenazione adeguata dei tessuti.
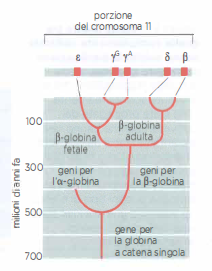
L’assetto α/β risale a un’antica duplicazione avvenuta agli inizi della radiazione dei vertebrati, verosimilmente circa 500 milioni di anni fa, seguita da divergenza delle due copie in linee distinte che codificano α-globine e β-globine. In seguito, ulteriori duplicazioni nel cluster “β-like” hanno dato origine a geni con specificità d’espressione temporale, inclusi geni espressi nella vita fetale (Figura 04.02-09). L’emoglobina fetale presenta una maggiore affinità per l’ossigeno rispetto alla forma adulta, facilitando il flusso di ossigeno dalla circolazione materna a quella fetale.
Duplicazioni successive hanno ampliato i cluster delle α- e delle β-globine in insiemi di geni con regolazione e caratteristiche funzionali differenziate. Mutazioni puntiformi che alterano le proprietà di legame all’ossigeno, insieme a variazioni nelle regioni regolative che modulano “quando” e “quanto” ciascun gene venga trascritto, hanno prodotto un repertorio di globine con profili di espressione stadio-specifici e differenti curve di saturazione. Accanto ai geni funzionali, i cluster ospitano numerosi pseudogeni, copie inattivate dall’accumulo di mutazioni deleterie, a testimonianza del fatto che molte duplicazioni non si traducono in nuove funzioni stabili. Sebbene qui si sia discusso il caso delle globine, percorsi analoghi hanno forgiato molte altre famiglie geniche del genoma umano.
Nei vertebrati, la presenza diffusa di famiglie geniche multiple suggerisce che oltre a duplicazioni locali si siano verificate duplicazioni su scala di intero genoma (WGD, whole-genome duplication). Evidenze comparative supportano l’ipotesi di due round antichi di WGD nelle prime fasi dell’evoluzione dei vertebrati, con la generazione transitoria di quattro copie per molti loci, poi in parte perse o riconfigurate. In alcuni cladi, in particolare nei pesci ossei, si riconosce un’ulteriore duplicazione, e in alcune linee (come salmonidi e carpe, cui appartiene anche il pesce zebra) eventi addizionali hanno aumentato la ridondanza genica fino a otto copie.
Ricostruire con precisione questi passaggi è complesso, poiché dopo una WGD intervengono ristrutturazioni, perdite differenziali di geni (“frazionamento”) e riassortimenti cromosomici. In alcuni organismi, però, la poliploidia è recente e chiaramente evidente: nel genere Xenopus coesistono specie strettamente affini con diverso contenuto di DNA, da forme diploidi a tetraploidi e ottaploidi. Tali salti di ploidia possono originare da mancata divisione cellulare dopo la replicazione nelle cellule germinali o dalla formazione di gameti non ridotti, seguiti da fecondazione.
Nelle piante coltivate la poliploidia è frequente e spesso vantaggiosa, perché può aumentare vigore, dimensioni dei frutti e adattabilità agronomica. In alcuni casi, come nell’uva apirena, la duplicazione del genoma comporta sterilità per alterata formazione dei semi. Mele, porri e patate sono tipicamente tetraploidi, mentre fragole e canna da zucchero mostrano livelli di ploidia più elevati, come l’ottaploidia (Figura 04.02-10).
Le WGD non producono solo ridondanza. Effetti di “dosage balance” favoriscono il mantenimento congiunto di reti di geni interagenti, mentre duplicati possono divergere condividendo le funzioni ancestrali o acquisendone di nuove. In sintesi, le WGD forniscono materiale grezzo per l’innovazione su scala di interi circuiti regolativi e vie metaboliche.
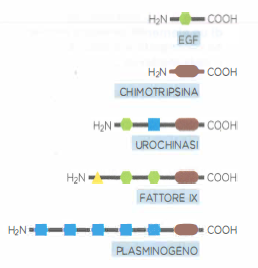
Molte proteine eucariotiche sono mosaici di domini funzionali. Tale modularità è riflessa nella struttura genica: sequenze codificanti (esoni) separate da ampi introni. Questa architettura facilita la ricombinazione all’interno degli introni e l’unione di esoni provenienti da geni distinti, un processo noto come rimescolamento degli esoni. La stessa logica di ricombinazione che genera duplicazioni (Figura 04.02-07) può, quando coinvolge introni omologhi di geni diversi, produrre geni ibridi con esoni interi provenienti da origini differenti.
Molte proteine attuali, costituite da insiemi di domini multicopia in combinazioni diverse, sono compatibili con un’origine per rimescolamento (Figura 04.02-11). A questa dinamica si aggiungono contributi degli elementi mobili, che possono mobilizzare moduli esonici o introdurre nuovi siti di splicing, e duplicazioni locali che replicano specifici domini. È plausibile che una parte significativa del proteoma umano derivi dalla ricombinazione e duplicazione ripetuta di un repertorio relativamente limitato di moduli esonici, ciascuno corrispondente a un dominio proteico compatto dell’ordine di poche decine-centinaia di amminoacidi, assemblati in configurazioni funzionali differenti.
Gli elementi genetici mobili sono sequenze in grado di spostarsi nel genoma e di proliferare, influenzandone architettura e funzione. Questi elementi, spesso parassitici, possono interrompere la funzione genica o modificarne la regolazione, e talvolta generare nuovi geni per fusione con segmenti preesistenti. Inserzioni in regioni codificanti causano mutazioni per frameshift o interruzione del reading frame; inserzioni in regioni regolative alterano pattern spazio-temporali di espressione (Figura 04.02-12). Alcune forme di emofilia sono state ricondotte a inserzioni che inattivano geni chiave coinvolti nella coagulazione.
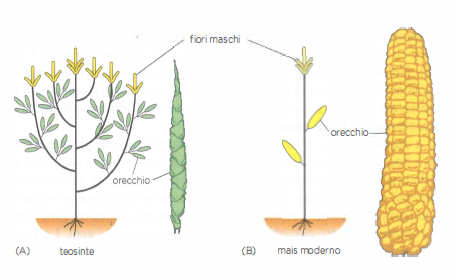
Molti elementi mobili trasportano siti riconosciuti da regolatori trascrizionali; la loro integrazione in prossimità di un gene può imporre un nuovo controllo trascrizionale e ridefinire reti regolative. Questi riassestamenti hanno avuto un ruolo rilevante nell’evoluzione delle piante domesticate. L’addomesticamento del mais a partire dal teosinte, per esempio, è stato favorito da poche variazioni genetiche di grande effetto, tra cui l’inserzione di un elemento mobile a monte di un gene implicato nello sviluppo del seme, che ha trasformato i piccoli semi duri del teosinte negli ampi chicchi del mais moderno (Figura 04.02-13).
Gli elementi mobili, oltre a inserire nuove sequenze, forniscono substrati di ricombinazione omologa non allelica disseminati nel genoma. Ectopiche ricombinazioni tra copie dello stesso elemento possono generare duplicazioni, delezioni e traslocazioni (Figura 04.02-07). È plausibile che alcune espansioni dei cluster della β-globina siano originate per ricombinazione tra elementi mobili abbondanti nel genoma umano. Dal punto di vista meccanicistico, si distinguono retrotrasposoni (classe I), che si muovono tramite RNA intermedio, e trasposoni a DNA (classe II), che si spostano direttamente come DNA; entrambi hanno lasciato un’impronta estesa nella nostra specie.
Oltre alle variazioni interne a un genoma, segmenti di DNA possono passare tra organismi non imparentati mediante trasferimento genico orizzontale (HGT). Negli eucarioti il fenomeno è raro, mentre nei batteri è comune e avviene tramite diversi meccanismi:
- coniugazione, passaggio diretto di DNA da cellula a cellula mediato da plasmidi o elementi integrativi (Figura 04.02-14);
- trasformazione, acquisizione di DNA libero dall’ambiente;
- trasduzione, trasferimento di geni per mezzo di batteriofagi.
Stime comparative indicano che Escherichia coli abbia acquisito una parte rilevante del proprio repertorio genico da altre specie batteriche negli ultimi cento milioni di anni. Oggi, l’HGT alimenta la rapida diffusione della resistenza agli antibiotici: geni che conferiscono resistenza possono muoversi tra specie su plasmidi, trasposoni e integroni, conferendo un vantaggio selettivo immediato. L’impatto clinico è evidente: numerosi antibiotici risultano meno efficaci contro patogeni un tempo sensibili. Un esempio emblematico è Neisseria gonorrhoeae, di cui la maggior parte dei ceppi ha acquisito resistenza alla penicillina, rendendo questo farmaco non più trattamento di prima scelta per la gonorrea.